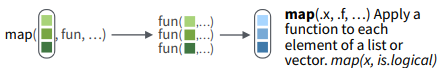
已经大半年没有更新博客了。。最近都跑去写分析报告半年没有R
这次记录下关于R循环(百万级以上)死慢死慢的问题,这个问题去年就碰到过,当时也尝试过多线程,but failed......昨天试了下,终于跑通了,而且过程还挺顺利
step1
先查下自己电脑几核的,n核貌似应该选跑n个线程,线程不是越多越好,线程个数和任务运行时间是条开口向下的抛物线,最高点预计在电脑的核数上。
detectCores( )检查当前电脑可用核数 我的是4所以step2选的是4
library(parallel) cl.cores <- detectCores()
step 2
多线程计算
setwd("C:\\Users\\siyuanmao\\Documents\\imdada\\0-渠道投放和新人券联动模型\\测算")
options(scipen=3) ##取消科学计数法
channel_ad_ios_data<-seq(0,50000,5000)
channel_ad_android_data<-seq(0,100000,10000)
library(parallel)
func <- function(n){#n=1
result_data<-read.csv("发券方案.csv",stringsAsFactors=FALSE)
total_coupon_solution_data<-read.csv("结果表框架.csv",stringsAsFactors=FALSE)
coupon_solution_data<-subset(result_data,solution== paste('方案',n,sep=""))
for (i in 1:11){#i=3
coupon_solution_data$channel_ad_cost[3]<-5000*(i-1)
for (j in 1:11){#j=5
coupon_solution_data$channel_ad_cost[4]<-10000*(j-1)
solution_mark<-paste('方案',n,i,j,sep="-")
coupon_solution_data$solution<-solution_mark
total_coupon_solution_data<-rbind(total_coupon_solution_data,coupon_solution_data)
}
}
print(solution_mark)
return(total_coupon_solution_data)
}
#func(10)
system.time({
x <- 1:7776
cl <- makeCluster(4) # 初始化四核心集群
results <- parLapply(cl,x,func) # lapply的并行版本
res.df <- do.call('rbind',results) # 整合结果
stopCluster(cl) # 关闭集群
})
df=as.data.frame(res.df)
原来非多线程的时候,我预计要跑12个小时以上,电脑发出呼呼~~的响声,查了下Python循环会快点,然后改为python版(已经很久没有用了,连个range都不会写,摸索了大半天才改好,但是速度还是慢==),于是改成多线程,运行25分钟就出结果了~~
补充:R语言 多线程
parallel包
包的安装
install.packages("parallel")
library(parallel)
包中常用函数
detectCores() 检查当前的可用核数
clusterExport() 配置当前环境
makeCluster() 分配核数
stopCluster() 关闭集群
parLapply() lapply()函数的并行版本
其实R语言本来就是一门向量化语言,如果是对于一个向量的操作,使用apply函数一族能获得比较高的效率,相比于for循环,这种高效来自于:
用C实现了for循环
减少对于data.frame等数据结构等不必要的拷贝
但是很多时候,如果想更快的话,光apply函数一族还不足够,这时候就能用上多线程。
R语言parallel包可以帮助实现多线程。
parLapply的简单代码实战
检查当前核数
cl.cores <- detectCores() #结果 > cl.cores [1] 8
启动集群和关闭集群
cl <- makeCluster(4) # 初始化四核心集群 ###并行任务 stopCluster(cl) # 关闭集群
parLapply执行多线程计算
#定义计算平方函数
square <- function(x)
{
return(x^2)
}
#利用并行计算计算平方函数
num <- c(1:3)
cl <- makeCluster(4) # 初始化四核心集群
results <- parLapply(cl,num,square)#调用parLapply并行计算平方函数
final <- do.call('c',results)#整合结果
stopCluster(cl) # 关闭集群
#结果
> final
[1] 1,4,9
思考:在如此小的计算方式下,开4个核计算是否比开一个核要快
答案:当然是不一定,因为涉及到调度方式等额外开销,所以不一定快,因为真正并行起作用的地方在于大数据量的计算。
时间开销对比
两段对比代码
#定义计算平方函数
square <- function(x)
{
#########
#一段冗余代码增加执行时间
y = 2*x
if(y <300)
{z = y}
else
{z = x}
##########
return(x^2)
}
num <- c(1:10000000)
#并行计算
print(system.time({
cl <- makeCluster(4) # 初始化四核心集群
results <- parLapply(cl,num,square)#调用parLapply并行计算平方函数
final <- do.call('c',results)#整合结果
stopCluster(cl) # 关闭集群
}))
#结果
用户 系统 流逝
7.89 0.27 19.01
#普通计算
print(system.time({
results <- lapply(num,square)
final <- do.call('c',results)#整合结果
}))
#结果
用户 系统 流逝
29.74 0.00 29.79
显然在数据量比较大的时候,并行计算的时间几乎就是于核数反比。不过,也不是多开几个核就好,注意内存很容易超支的,每个核都分配相应的内存,所以要注意内存开销。出现内存问题的时候,需要检查是否代码是否合理,R语言版本(64位会比32位分配的内存大),核分配是否合理。
上一级环境中变量的引入
R语言里边对于环境变量有着有趣的定义,一层套一层,这里不做深入展开。
类似于在c语言函数中使用全局变量,R在执行并行计算的时候,如果需要计算的函数出现在全局(上一级),那么就需要声明引入这个变量,否则将会报错。
#定义计算幂函数
base = 2
square <- function(x)
{
return(x^base)
}
num <- c(1:1000000)
#利用并行计算计算幂函数
cl <- makeCluster(4) # 初始化四核心集群
results <- parLapply(cl,num,square)#调用parLapply并行计算平方函数
final <- do.call('c',results)#整合结果
stopCluster(cl) # 关闭集群
#结果报错
Error in checkForRemoteErrors(val) :
4 nodes produced errors; first error: 找不到对象'base'
#利用并行计算计算幂函数
cl <- makeCluster(4) # 初始化四核心集群
clusterExport(cl,"base",envir = environment())
results <- parLapply(cl,num,square)#调用parLapply并行计算平方函数
final <- do.call('c',results)#整合结果
stopCluster(cl) # 关闭集群
#结果
> final
[1] 1,4,9,16,25.......
foreach包
除了parallel包以外,还有针对并行for循环的foreach包,foreach()的使用也与parLapply()类似,两个功能也类似,其中遇到的问题也类似。
包的安装
install.packages("foreach")
library(parallel)
foreach的使用
#定义计算幂函数
square <- function(x)
{
return(x^2)
}
非并行情况的使用:
参数中的combine就是整合结果的函数,可以是c,可以是rbind,也可以是+等
results = foreach(x = c(1:3),.combine = 'c') %do% square(x) #结果 > results [1] 1,4,9
并行情况的使用:
注意并行情况的时候,需要与parallel包进行配合,引入library(doParallel)。同时%do%需要改成%dopar%。另外与parallel包不一样的是,需要多加一句registerDoParallel(cl)来注册核进行使用。
cl <- makeCluster(4) registerDoParallel(cl) results = foreach(x = c(1:100000),.combine = 'c') %dopar% square(x) stopCluster(cl)
上一级环境中变量的引入
同parallel包并行计算前需要clusterExport()来引入全局变量一样,foreach也同样需要声明,不同的是,foreach声明方式直接写在foreach()的参数export里边。
#定义计算幂函数
base = 2
square <- function(x)
{
return(x^base)
}
cl <- makeCluster(4)
registerDoParallel(cl)
results = foreach(x = c(1:100000),.combine = 'c',.export ='base' ) %dopar% square(x)
stopCluster(cl)
以上为个人经验,希望能给大家一个参考,也希望大家多多支持好代码网。如有错误或未考虑完全的地方,望不吝赐教。